Structural search and advanced query search is temporarily unavailable. We are working to fix this issue. Thank you for your support and patience.
L-Alanine-L-glutamate (M2MDB001650)
| Record Information | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Version | 2.0 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Creation Date | 2012-07-30 14:55:05 -0600 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Update Date | 2015-06-03 17:20:59 -0600 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Secondary Accession Numbers |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Identification | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
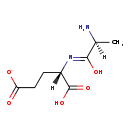
| Name: | L-Alanine-L-glutamate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Description | L-Alanine-L-glutamate is a dipeptide composed of alanine and glutamate. It is an incomplete breakdown product of protein digestion or protein catabolism. Some dipeptides are known to have physiological or cell-signaling effects although most are simply short-lived intermediates on their way to specific amino acid degradation pathways following further proteolysis. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Structure | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Synonyms: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Chemical Formula: | C8H13N2O5 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Weight: | Average: 217.1992 Monoisotopic: 217.082446536 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| InChI Key: | VYZAGTDAHUIRQA-WHFBIAKZSA-M | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| InChI: | InChI=1S/C8H14N2O5/c1-4(9)7(13)10-5(8(14)15)2-3-6(11)12/h4-5H,2-3,9H2,1H3,(H,10,13)(H,11,12)(H,14,15)/p-1/t4-,5-/m0/s1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CAS number: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| IUPAC Name: | (4S)-4-{[(2S)-2-amino-1-hydroxypropylidene]amino}-4-carboxybutanoate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Traditional IUPAC Name: | (4S)-4-{[(2S)-2-amino-1-hydroxypropylidene]amino}-4-carboxybutanoate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| SMILES: | [H][C@@](C)(N)C(O)=N[C@@]([H])(CCC([O-])=O)C(O)=O | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Chemical Taxonomy | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Description | belongs to the class of organic compounds known as dipeptides. These are organic compounds containing a sequence of exactly two alpha-amino acids joined by a peptide bond. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Kingdom | Organic compounds | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Super Class | Organic acids and derivatives | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Class | Carboxylic acids and derivatives | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Sub Class | Amino acids, peptides, and analogues | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Direct Parent | Dipeptides | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Alternative Parents |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Substituents |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Molecular Framework | Aliphatic acyclic compounds | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| External Descriptors |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Physical Properties | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| State: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Charge: | -2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Melting point: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Experimental Properties: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Predicted Properties |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Biological Properties | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Cellular Locations: | Cytoplasm | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reactions: | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| SMPDB Pathways: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| KEGG Pathways: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| EcoCyc Pathways: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Concentrations | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Not Available | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Spectra | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Spectra: | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| References | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| References: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Synthesis Reference: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Material Safety Data Sheet (MSDS) | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Links | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| External Links: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Enzymes
- General function:
- Involved in hydrolase activity
- Specific function:
- Dipeptidase with broad substrate specificity. Requires dipeptide substrates with an unblocked N-terminus and the amino group in the alpha or beta position. Non-protein amino acids and proline are not accepted in the C-terminal position, whereas some dipeptide amides and formyl amino acids are hydrolyzed. Also shows cysteinylglycinase activity, which is sufficient for E.coli to utilize cysteinylglycine as a cysteine source
- Gene Name:
- pepD
- Uniprot ID:
- P15288
- Molecular weight:
- 52915
Reactions
| Hydrolysis of dipeptides, preferentially hydrophobic dipeptides including prolyl amino acids. |
- General function:
- Involved in cellular amino acid catabolic process
- Specific function:
- Catalyzes the epimerization of L-Ala-D-Glu to L-Ala-L- Glu and has a role in the recycling of the murein peptide, of which L-Ala-D-Glu is a component. Is also able to catalyze the reverse reaction and the epimerization of all the L-Ala-X dipeptides, except L-Ala-L-Arg, L-Ala-L-Lys and L-Ala-L-Pro. Is also active with L-Gly-L-Glu, L-Phe-L-Glu, and L-Ser-L-Glu, but not with L-Glu-L-Glu, L-Lys-L-Glu, L-Pro-L-Glu, L-Lys-L-Ala, or D- Ala-D-Ala
- Gene Name:
- ycjG
- Uniprot ID:
- P51981
- Molecular weight:
- 34674
Reactions
| L-alanyl-D-glutamate = L-alanyl-L-glutamate. |
Transporters
- General function:
- Involved in peptide transporter activity
- Specific function:
- Proton-dependent permease that transports di- and tripeptides. Has a clear preference for dipeptides and tripeptides composed of L-amino acids, and discriminates dipeptides on the basis of the position of charges within the substrate
- Gene Name:
- dtpB
- Uniprot ID:
- P36837
- Molecular weight:
- 53575
- General function:
- Involved in peptide transporter activity
- Specific function:
- Probable proton-dependent permease that transports di- and tripeptides. Shows significantly higher specificity towards dipeptides
- Gene Name:
- yjdL
- Uniprot ID:
- P39276
- Molecular weight:
- 53054
- General function:
- Involved in peptide transporter activity
- Specific function:
- Probable proton-dependent permease that transports dipeptides
- Gene Name:
- dtpD
- Uniprot ID:
- P75742
- Molecular weight:
- 54158
- General function:
- Involved in peptide transporter activity
- Specific function:
- Proton-dependent permease that transports di- and tripeptides as well as structurally related peptidomimetics such as aminocephalosporins into the cell. Has a clear preference for dipeptides and tripeptides composed of L-amino acids, and discriminates dipeptides on the basis of the position of charges within the substrate
- Gene Name:
- dtpA
- Uniprot ID:
- P77304
- Molecular weight:
- 53991
- General function:
- Involved in transporter activity
- Specific function:
- Non-specific porin
- Gene Name:
- ompN
- Uniprot ID:
- P77747
- Molecular weight:
- 41220
- General function:
- Involved in transporter activity
- Specific function:
- Uptake of inorganic phosphate, phosphorylated compounds, and some other negatively charged solutes
- Gene Name:
- phoE
- Uniprot ID:
- P02932
- Molecular weight:
- 38922
- General function:
- Involved in transporter activity
- Specific function:
- OmpF is a porin that forms passive diffusion pores which allow small molecular weight hydrophilic materials across the outer membrane. It is also a receptor for the bacteriophage T2
- Gene Name:
- ompF
- Uniprot ID:
- P02931
- Molecular weight:
- 39333
- General function:
- Involved in transporter activity
- Specific function:
- Forms passive diffusion pores which allow small molecular weight hydrophilic materials across the outer membrane
- Gene Name:
- ompC
- Uniprot ID:
- P06996
- Molecular weight:
- 40368