Structural search and advanced query search is temporarily unavailable. We are working to fix this issue. Thank you for your support and patience.
UDP-N-Acetylmuramoyl-L-alanine (M2MDB000648)
| Record Information | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Version | 2.0 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Creation Date | 2012-05-31 14:07:09 -0600 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Update Date | 2015-06-03 15:54:49 -0600 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Secondary Accession Numbers |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Identification | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Name: | UDP-N-Acetylmuramoyl-L-alanine | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Description | Udp-n-acetylmuramoyl-l-alanine belongs to the class of Pyrimidine Nucleotide Sugars. These are pyrimidine nucleotides bound to a saccharide derivative through the terminal phosphate group. (inferred from compound structure)UDP-N-acetylmuramoyl-L-alanine (UDP-MurNAc-L-Ala), which is the nucleotide substrate of the D-glutamic-acid-adding enzyme (the murD gene product) catalyzing the subsequent step in the pathway for peptidoglycan synthesis, appears to be an effector of the racemase activity. (PMID 8098327) MurD (UDP-N-acetylmuramoyl-L-alanine:D-glutamate ligase) is the second enzyme in the series of Mur ligases, and it catalyzes the addition of D-glutamic acid (D-Glu) to the cytoplasmic intermediate UDP-N-acetylmuramoyl-L-alanine (UMA). (PMID 19007109) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
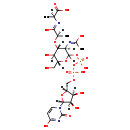
| Structure | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Synonyms: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Chemical Formula: | C23H36N4O20P2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Weight: | Average: 750.4943 Monoisotopic: 750.139812636 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| InChI Key: | NTMMCWJNQNKACG-CPDMUANVSA-N | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| InChI: | InChI=1S/C23H36N4O20P2/c1-8(21(35)36)24-19(34)9(2)43-18-14(25-10(3)29)22(45-11(6-28)16(18)32)46-49(40,41)47-48(38,39)42-7-12-15(31)17(33)20(44-12)27-5-4-13(30)26-23(27)37/h4-5,8-9,11-12,14-18,20,22,28,31-33H,6-7H2,1-3H3,(H,24,34)(H,25,29)(H,35,36)(H,38,39)(H,40,41)(H,26,30,37)/t8-,9+,11+,12+,14+,15+,16+,17+,18+,20+,22?/m0/s1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CAS number: | 78738-65-5 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| IUPAC Name: | (2S)-2-{[(2R)-2-{[(3R,4R,5S,6R)-2-({[({[(2R,3S,4R,5R)-3,4-dihydroxy-5-(4-hydroxy-2-oxo-1,2-dihydropyrimidin-1-yl)oxolan-2-yl]methoxy}(hydroxy)phosphoryl)oxy](hydroxy)phosphoryl}oxy)-5-hydroxy-3-[(1-hydroxyethylidene)amino]-6-(hydroxymethyl)oxan-4-yl]oxy}-1-hydroxypropylidene]amino}propanoic acid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Traditional IUPAC Name: | (2S)-2-{[(2R)-2-{[(3R,4R,5S,6R)-2-[({[(2R,3S,4R,5R)-3,4-dihydroxy-5-(4-hydroxy-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy(hydroxy)phosphoryl}oxy(hydroxy)phosphoryl)oxy]-5-hydroxy-3-[(1-hydroxyethylidene)amino]-6-(hydroxymethyl)oxan-4-yl]oxy}-1-hydroxypropylidene]amino}propanoic acid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| SMILES: | [H][C@](C)(O[C@@]1([H])[C@]([H])(O)[C@@]([H])(CO)OC([H])(OP(O)(=O)OP(O)(=O)OC[C@@]2([H])O[C@@]([H])(N3C=CC(O)=NC3=O)[C@]([H])(O)[C@]2([H])O)[C@]1([H])N=C(C)O)C(O)=N[C@@]([H])(C)C(O)=O | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Chemical Taxonomy | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Description | belongs to the class of organic compounds known as pyrimidine nucleotide sugars. These are pyrimidine nucleotides bound to a saccharide derivative through the terminal phosphate group. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Kingdom | Organic compounds | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Super Class | Nucleosides, nucleotides, and analogues | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Class | Pyrimidine nucleotides | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Sub Class | Pyrimidine nucleotide sugars | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Direct Parent | Pyrimidine nucleotide sugars | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Alternative Parents |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Substituents |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Molecular Framework | Aromatic heteromonocyclic compounds | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| External Descriptors |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Physical Properties | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| State: | Solid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Charge: | -2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Melting point: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Experimental Properties: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Predicted Properties |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Biological Properties | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Cellular Locations: | Cytoplasm | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reactions: | Adenosine triphosphate + D-Glutamic acid + UDP-N-Acetylmuramoyl-L-alanine <> ADP + Hydrogen ion + Phosphate + UDP-N-Acetylmuramoyl-L-alanyl-D-glutamate L-Alanine + Adenosine triphosphate + UDP-N-Acetylmuraminate <> ADP + Hydrogen ion + Phosphate + UDP-N-Acetylmuramoyl-L-alanine Adenosine triphosphate + UDP-N-Acetylmuramoyl-L-alanine + D-Glutamic acid <> ADP + Phosphate + UDP-N-Acetylmuramoyl-L-alanyl-D-glutamate Adenosine triphosphate + UDP-N-Acetylmuraminate + L-Alanine <> ADP + Phosphate + UDP-N-Acetylmuramoyl-L-alanine D-Glutamic acid + UDP-N-Acetylmuramoyl-L-alanine + Adenosine triphosphate > Hydrogen ion + UDP-N-Acetylmuramoyl-L-alanyl-D-glutamate + Phosphate + ADP Adenosine triphosphate + UDP-N-Acetylmuraminate + L-Alanine > ADP + Inorganic phosphate + UDP-N-Acetylmuramoyl-L-alanine Adenosine triphosphate + UDP-N-Acetylmuramoyl-L-alanine + DL-Glutamic acid > ADP + Inorganic phosphate + UDP-N-Acetylmuramoyl-L-alanyl-D-glutamate UDP-N-acetylmuraminate + Adenosine triphosphate + L-Alanine + UDP-N-Acetylmuraminate + L-Alanine > UDP-N-Acetylmuramoyl-L-alanine + Adenosine diphosphate + Phosphate + ADP UDP-N-Acetylmuramoyl-L-alanine + Adenosine triphosphate + D-Glutamic acid > Adenosine diphosphate + Phosphate + UDP-N-acetylmuramoyl-L-alanyl-D-glutamate + ADP + UDP-N-Acetylmuramoyl-L-alanyl-D-glutamate UDP-N-acetyl-α-D-muramate + L-Alanine + Adenosine triphosphate + L-Alanine > Adenosine diphosphate + Phosphate + Hydrogen ion + UDP-N-Acetylmuramoyl-L-alanine + ADP L-Alanine + Adenosine triphosphate + UDP-N-Acetylmuraminate <> ADP + Hydrogen ion + Phosphate + UDP-N-Acetylmuramoyl-L-alanine Adenosine triphosphate + D-Glutamic acid + UDP-N-Acetylmuramoyl-L-alanine <> ADP + Hydrogen ion + Phosphate + UDP-N-Acetylmuramoyl-L-alanyl-D-glutamate L-Alanine + Adenosine triphosphate + UDP-N-Acetylmuraminate <> ADP + Hydrogen ion + Phosphate + UDP-N-Acetylmuramoyl-L-alanine Adenosine triphosphate + D-Glutamic acid + UDP-N-Acetylmuramoyl-L-alanine <> ADP + Hydrogen ion + Phosphate + UDP-N-Acetylmuramoyl-L-alanyl-D-glutamate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| SMPDB Pathways: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| KEGG Pathways: | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| EcoCyc Pathways: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Concentrations | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Not Available | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Spectra | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Spectra: | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| References | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| References: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Synthesis Reference: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Material Safety Data Sheet (MSDS) | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Links | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| External Links: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Enzymes
- General function:
- Involved in ATP binding
- Specific function:
- Cell wall formation. Catalyzes the addition of glutamate to the nucleotide precursor UDP-N-acetylmuramoyl-L-alanine (UMA)
- Gene Name:
- murD
- Uniprot ID:
- P14900
- Molecular weight:
- 46973
Reactions
| ATP + UDP-N-acetylmuramoyl-L-alanine + glutamate = ADP + phosphate + UDP-N-acetylmuramoyl-L-alanyl-D-glutamate. |
- General function:
- Involved in ATP binding
- Specific function:
- Cell wall formation
- Gene Name:
- murC
- Uniprot ID:
- P17952
- Molecular weight:
- 53626
Reactions
| ATP + UDP-N-acetylmuramate + L-alanine = ADP + phosphate + UDP-N-acetylmuramoyl-L-alanine. |